[Bruker NewsLetter]Bruker升級影像質譜、代謝體學、生物相似藥等解決方案
Bruker升級影像質譜、代謝體學、生物相似藥等解決方案
影像軟體SCiLS整合MALDI-2資料庫與QuPath平台
今年ASMS 2020會議上,Bruker推出了世界上第一個商用MALDI後電離(Post-Ionization,PI)離子源MALDI-2 (圖1),可用於timsTOF fleX質譜儀上,全新的MALDI-2離子源可將小分子和脂質分析的靈敏度提高一到兩個數量級,從而進一步擴大MALDI技術的質譜成像應用範圍。在對應的軟體上,影像處理軟體SCiLS的最新版本也整合了MALDI-2資料庫,並能與MetaboScape® 2021跨軟體平台整合,讓代謝體學的研究成為一完整解決方案,與MetaboScape® 的SpatialOMx工作流程集成。

圖1 Bruker發表MALDI-2:在MALDI游離源後使用第二道雷射激發中性分子提升游離效率
測試MALDI-2對添加BI-YYY藥物標準品在組織切片上靈敏度的增強,並在大鼠用Chloroquine或BI-YYY給藥後,用MALDI-2成像分析,以確定靈敏度提高是否轉變為重要的藥物定位資訊。此外,還研究了Chloroquine代謝物分佈,大鼠在分別給予Chloroquine或BI-YYY藥物2小時或24小時後取組織樣本,在配置了MALDI-2的timsTOF fleX質譜系統上採集切片的質譜資料,圖原尺寸為50µm。用SCiLS軟體處理得到組織切片分子圖像,然後用MetaboScape® 2021軟體中全新的BioTransformer模組,預測了35個代謝產物1,對其中19個特別代謝物做了鑑定和強度測量,並計算了相對含量。小分子藥物游離效率因使用MALDI-2顯著提高了所有受測試化合物的靈敏度,其中新的藥物BI-YYY的峰值強度使用MALDI-2提高了300倍(圖2),結果皆能展示在SCiLS軟體中。
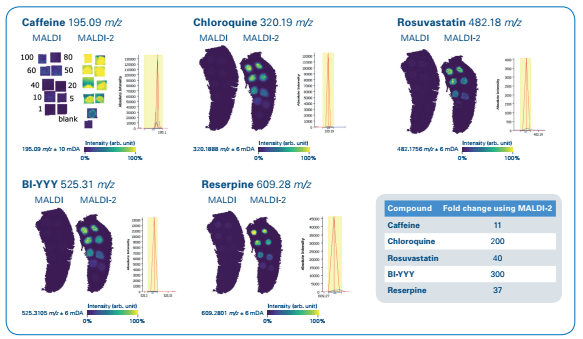
圖2 使用MALDI-2後,小分子化合物的靈敏度得到數量級的提高
此外,影像軟體SCiLS新增QuPath型態分析能力,QuPath是一個開源性、可擴展的軟體平台,它提供了廣泛的功能,包括:標註和視覺化工具;IHC和H&E分析的工作流程;常見的新穎演算法,例如: 細胞分割、組織微陣列解排列;互動式機器學習,例如:細胞和紋理分類;基於物件的分層資料模型,添加新功能或對不同圖像源支援,易於與其他工具集成,例如MATLAB和ImageJ可用於整個切片圖像分析。QuPath一般為病理學家廣泛使用。Bruker現在發展出在QuPath中標註的區域可通過SCiLS外掛程式導出,外掛程式將圖像和標註導出到SCiLS ,即可將帶有標註的圖像載入SCiLS檔中,因此新版的SCiLS提供更佳的標註工具,支援多種組織學的數據格式(圖3),足見Bruker整合臨床應用與基礎研究的發展方向。
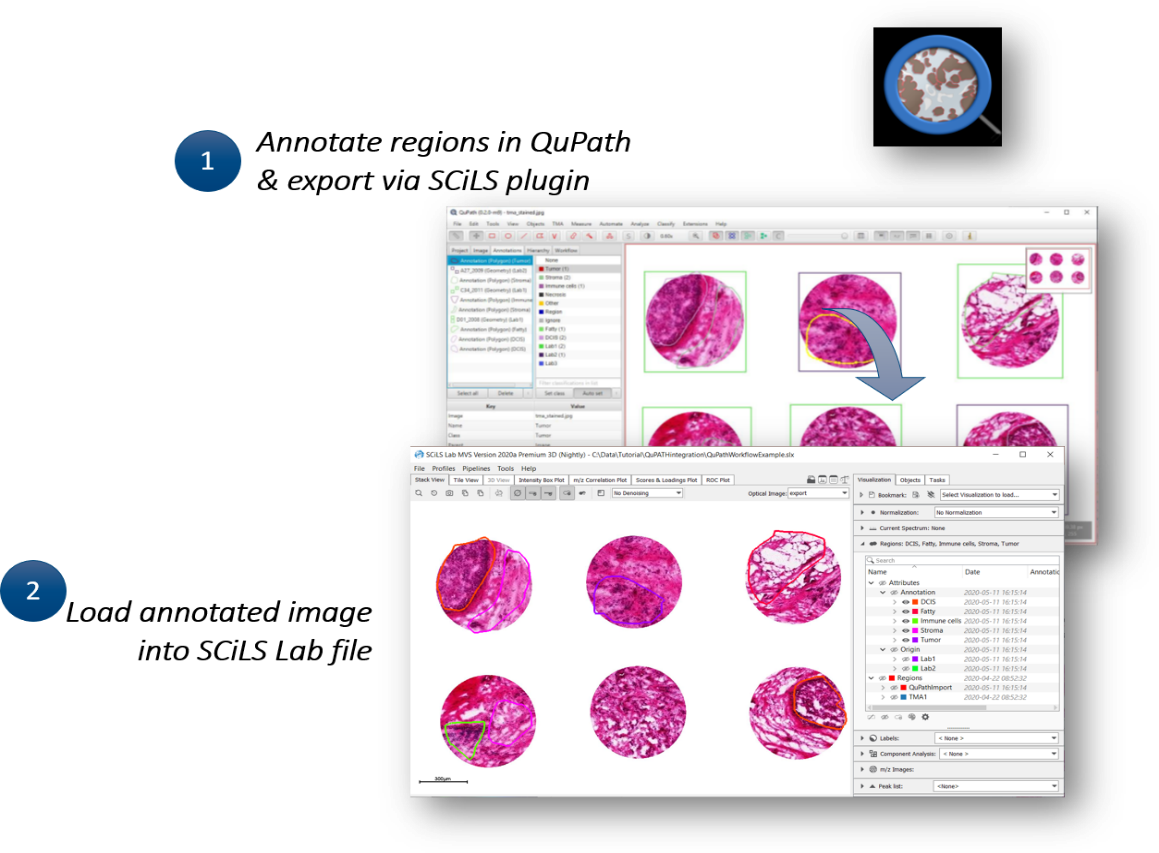
圖3 QuPath與SCiLS軟體平台整合示意圖
因應Bruker推出的timsTOF fleX,SCiLS的新功能也包括獨立呈現TIMS模式下的數據可視化,亦即通過CCS分離圖像,為MALDI分子組織圖像提供特徵(代謝物、脂質、聚醣等)的獨特CCS標註。以圖4為例,此為老鼠組織成像,m/z同為754.612的化合物,其中有兩種不同的CCS值分子 (橘色顯示1/K0為1.36的分佈,藍色顯示1/K0為1.42的分佈)。
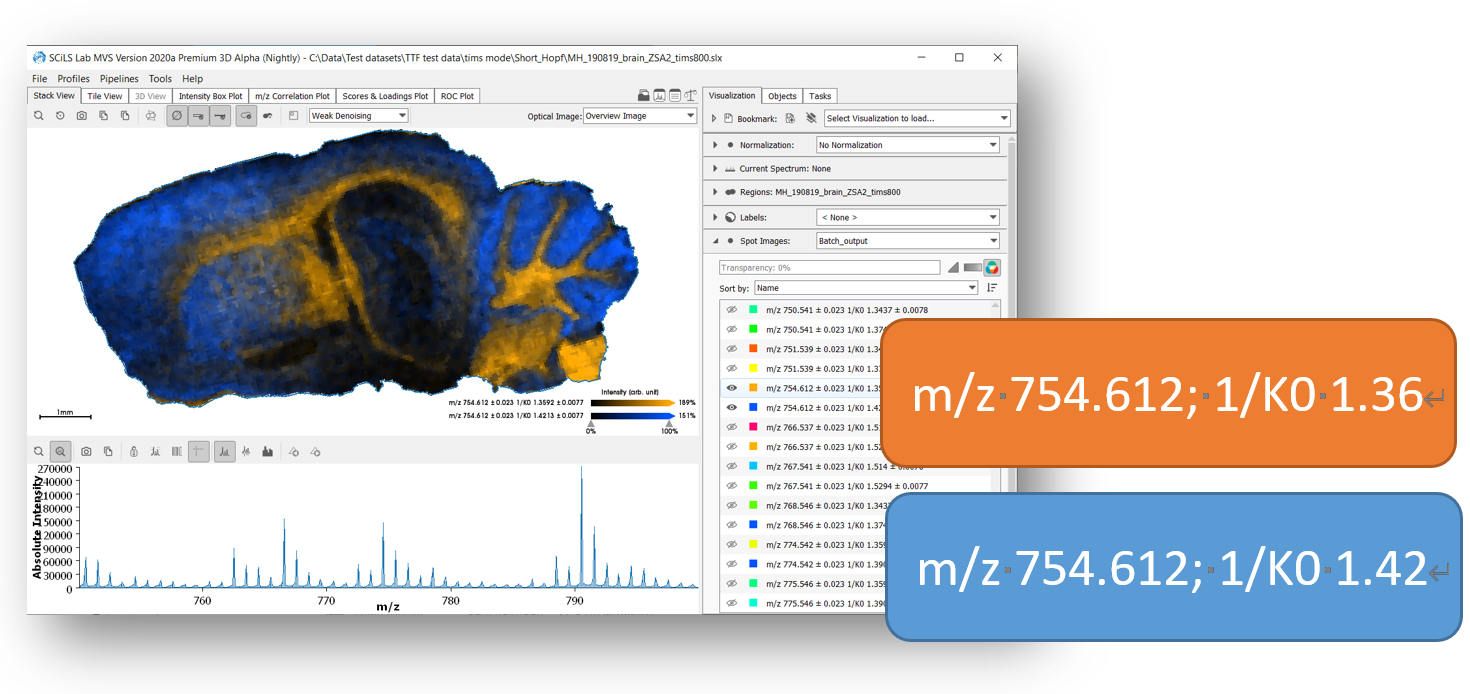
圖4 相同m/z的化合物,在CCS離子遷移率分離維度上,呈現的訊號和影像
MetaboScape®在表型與代謝體學上應用新模組BioTransformer
非標的代謝體學涉及不同的研究領域,如藥物開發、表型組學、環境暴露和非標的篩選等。雖然研究目標不盡相同,但卻有相同的需求,如: 差異標誌物的尋找、未知化合物鑑定和化合物相對定量等。LC-PASEF工作流程結合MetaboScape®軟體,以4D特徵提取器(T-ReX 4D)從timsTOF資料中提取CCS值,提高非標的物鑑定信心度。整體化解決方案包括批量資料的採集處理、統計學分析、化合物鑑定、數據可視化以及代謝途徑解析。無論是使用ESI或MALDI影像數據都可進行藥物代謝研究,新版MetaboScape® 2021增加了新模組BioTransformer,提供代謝時間序列分析和化合物代謝物預測的功能。
以不同反應終止時間(0、5、15、30、45、60、90和120 min)的藥物體外代謝肝微粒體模型為範例,在時間序列下依序終止反應收集產物,所有樣品經離心稀釋後使用Bruker Elute UPLC-timsTOF Pro進行分析(+ESI, PASEF模式),再將數據導入MetaboScape® 2021進行資料分析。在MetaboScape軟體中可使用SmartFormula、Analyte List、Spectral Library、CCS predict和BioTransformer等工具對化合物進行鑑定,Chlorpromazine體外代謝肝微粒體模型的代謝物模擬結果見圖5。
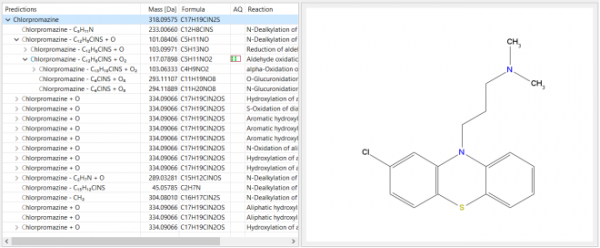
圖5 使用MetaboScape預測的Chlorpromazine代謝物列表
此預測結果直接連結到資料庫中進行比對,以Chlorpromazine+ O(C17H19ClN2SO,m/z 335.0979)代謝物為例,在資料庫中共比對到兩個可能結果,其層析滯留時間分別為2.72min和2.81min(圖6,綠色與紫色EIC結果)。
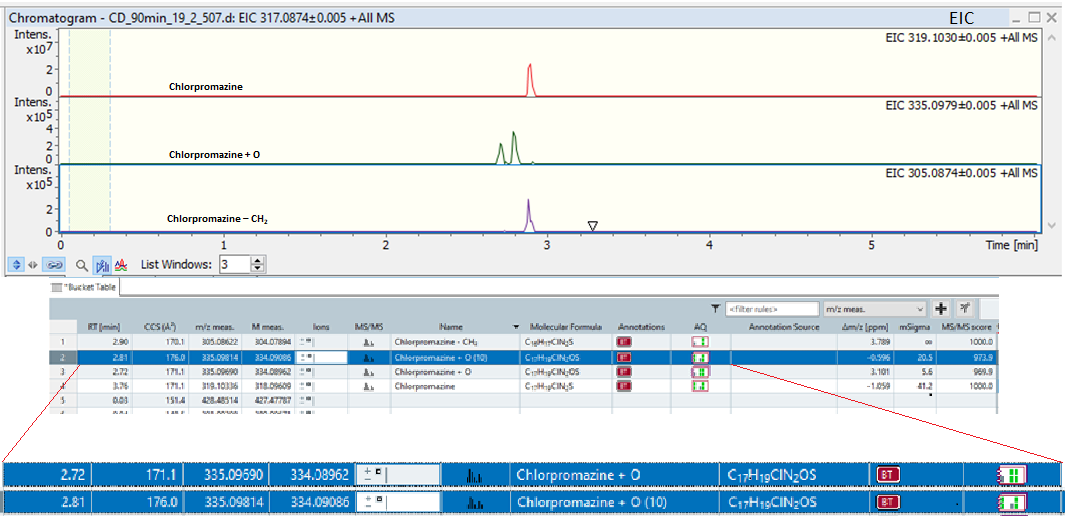
圖6 MetaboScape比對Chlorpromazine+ O代謝物的結果
基於文獻中Chlorpromazine+O代謝物可能存在同分異構體(hydroxy-,sulfoxide和N-oxide結構)的情況,對資料提取離子遷移CCS圖,。離子遷移分離維度圖顯示2.72min和2.81 min的CCS值分別為171.2和175.9(圖7),顯示分別為兩種不同的代謝物。結合代謝物MS/MS資料庫(MetaboBASE® Personal Library 3.0)的比對結果,保留時間為2.72min的化合物sulfoxide結構的與資料庫中MS/MS偏差較小,而另一可能代謝物N-oxide結構的二級質譜圖匹配度很低。再進一步對比實測和預測的CCS值,sulfoxide結構的偏差為1.3%,hydroxy-結構的偏差為-0.9 %,故2.72min化合物為Chlorpromazine-SO,同理,推測得2.81min代謝物為Chlorpromazine-7OH,MS/MS與CCS的雙重驗證提高非標的物鑑定可信度。

圖7 離子遷移分離圖顯示2.72分鐘和2.81分鐘的CCS值分別為171.2和175.9 Å2
MetaboScape® 2021也可以提供代謝物隨時間變化的視覺化結果。圖8左為滯留時間為2.72min的Chlorpromazine-SO,右為滯留時間為2.81min的Chlorpromazine-7OH 兩種代謝物強度隨時間變化的情況。可以發現Chlorpromazine-SO代謝物的體外肝微粒體代謝狀況持續增長到120分鐘後,而Chlorpromazine-7OH則是在90分鐘後開始衰減。
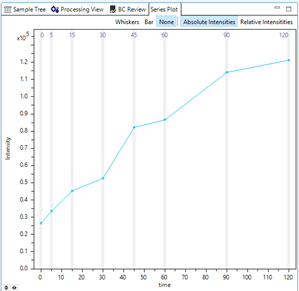
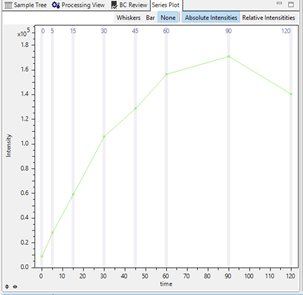
圖8 (左) RT 2.72min Chlorpromazine-SO (右) RT 2.81min Chlorpromazine-7OH代謝物隨時間強度變化
BioPharma Compass®: 加速生物製劑與生物相似藥開發與分析
美國的前10大暢銷藥物中有5種為單株抗體藥物,也有數據指出生物製劑佔美國處方藥的40%2, 3。面對此趨勢,生物製劑與生物相似藥開發過程需要新技術來增加對產品與製程的瞭解、品質控管,才能加速藥品開發過程。這些趨勢與美國食品藥物管理局(FDA)所提出的品質源自設計(QbD)理念相符,旨在提升藥物品質,減少不良事件。生物製藥產業因此面臨更多挑戰,複雜度高的大分子抗體或是抗體-藥物接合物 (Antibody-drug conjugate),通常需要精密與多樣分析工具才能鑑定,並且必須是靈敏的監測工具才能在開發與製程中掌握這些大分子沒有發生改變,而影響後續的藥效。
質譜(MS)分析技術與日俱進,已成為蛋白質類藥物分析最佳工具,利用MS進行多種屬性監測(Multi-Attribute Methods,以下簡稱MAM)的趨勢也隨之提高,原因在於MS的靈敏度與選擇性比傳統光學或物理化學偵測更佳,LC-MS更能在單次分析中監測與定量多種產品關鍵品質屬性(Critical quality attributes,簡稱CQA),此外,數據與文件的產出需有符合法規要求的資訊系統與平台搭配,以確保資料完整性,並可轉換到各製造階段與對應的法規需求,因此軟體的整合更是工作流程的主導角色。
Bruker的BioPharma Compass®提供自動化的生物藥物MAM分析程序,提供了從自動測量到報告生成的專用LC和MS工作流程,例如: 驗證蛋白質序列並量化產物和過程相關的異質性,例如:修飾圖譜與定量結果。這些MAM能使用高分辨率ESI-QTOF或MALDI-TOF質譜得到數據後,整合至BioPharma Compass®中,並自動產出符合法規的報告,減少人員與報告的處理時間。
新版的BioPharma Compass® 2021除了維持先前版本的完整蛋白質分析(例如:醣型和異質性篩選)、Top-down的蛋白質序列變異測序與定位、胜肽段平行序列分析和屬性監控、LC-ESI QTOF和MALDI-TOF的多重目標物篩選(例如: 寡核苷酸和胜肽合成品質控制),增加了使用PASEF數據和Mascot進行宿主細胞蛋白(Host cell proteins,簡稱HCP)識別、蛋白質篩選擴展出平均藥物抗體比(average drug-antibody ratioru,簡稱DAR)4計算與蛋白質裁截分析(Clipping analysis)、Top-down測序ESI / MALDI工作流程也可用於Clipping analysis。
圖9為BioPharma Compass®針對完整蛋白質分析篩選結果,利用軟體內建的向量式演算法為結果評分,得到直觀的分析結果(圖中綠色或紅色標註)。
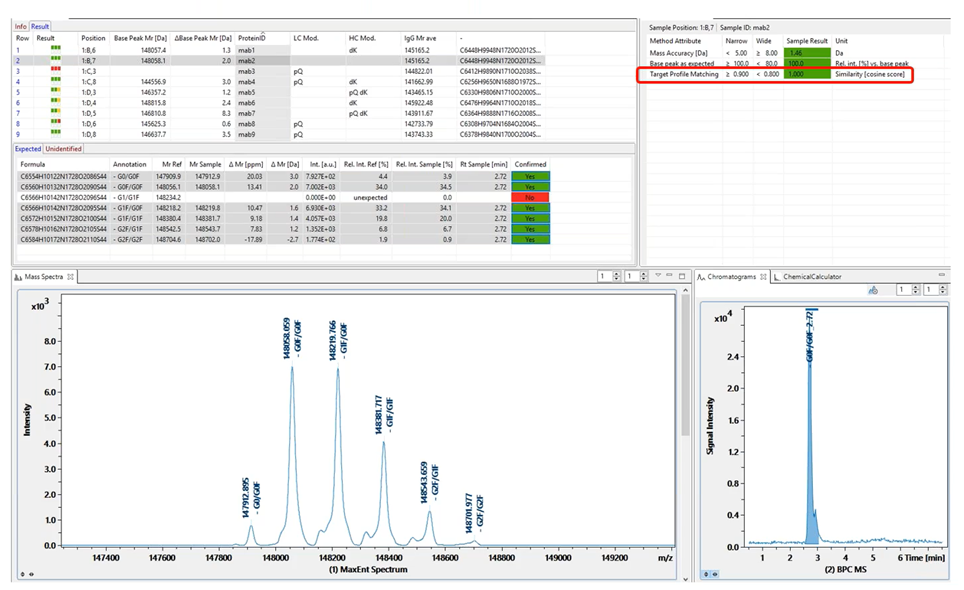
圖9 完整蛋白質異質性篩選結果展示
圖10展示ADC藥物Kadcyla(Trastuzumab+Emtansine)的LC-ESI QTOF分析後載入BioPharma Compass®計算出的平均DAR結果為3.49,以及DAR分佈從0到7,蛋白質篩選自動化工作流程簡化平均DAR計算,建立複雜的目標參考資料。
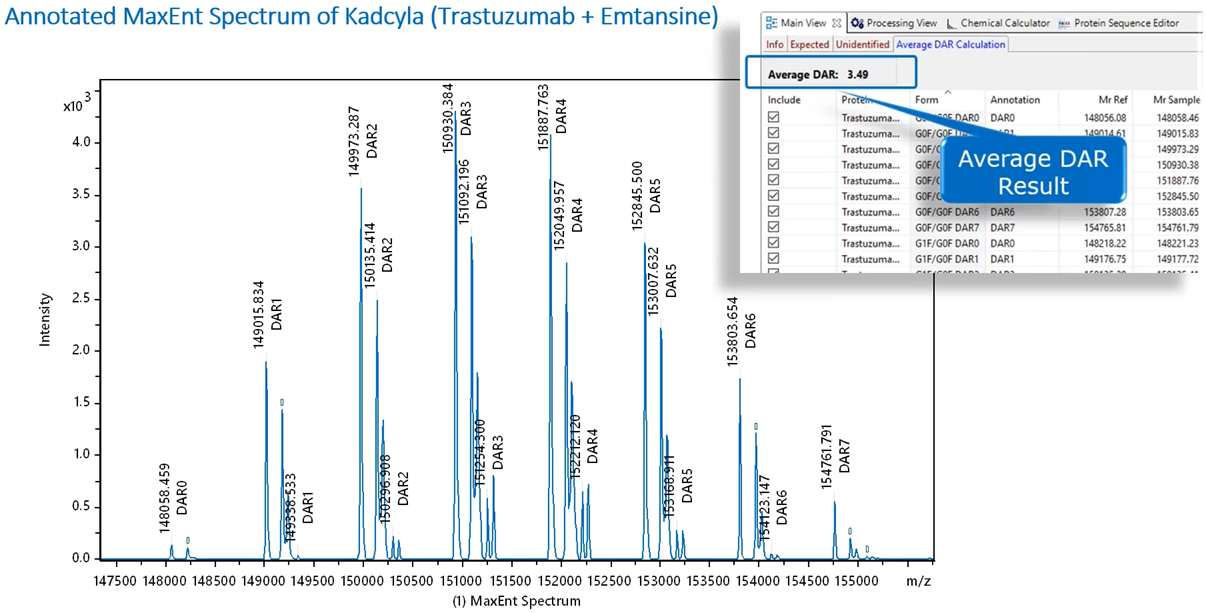
圖10 Kadcyla帶標註的DAR分布以及平均計算結果
在生產或製造抗體藥的過程中,由於步驟繁瑣,易發生抗體藥物異質性(Heterogeneity),或是裁截(clipping)現象,伴隨副產物產生,BioPharma Compass®也提供了自動化的clipping分析結果。圖11為Panitumumab的Clipping分析結果。FabULOUS(SpeB)是一種重組產生的半胱氨酸蛋白酶,在還原條件下可在許多物種的抗體IgG鉸鏈區中裂解,由於此酵素裂解IgG1特異性很高,但是對於IgG2的切點特異性卻較低,明顯可見Panitumumab的clipping副產物。
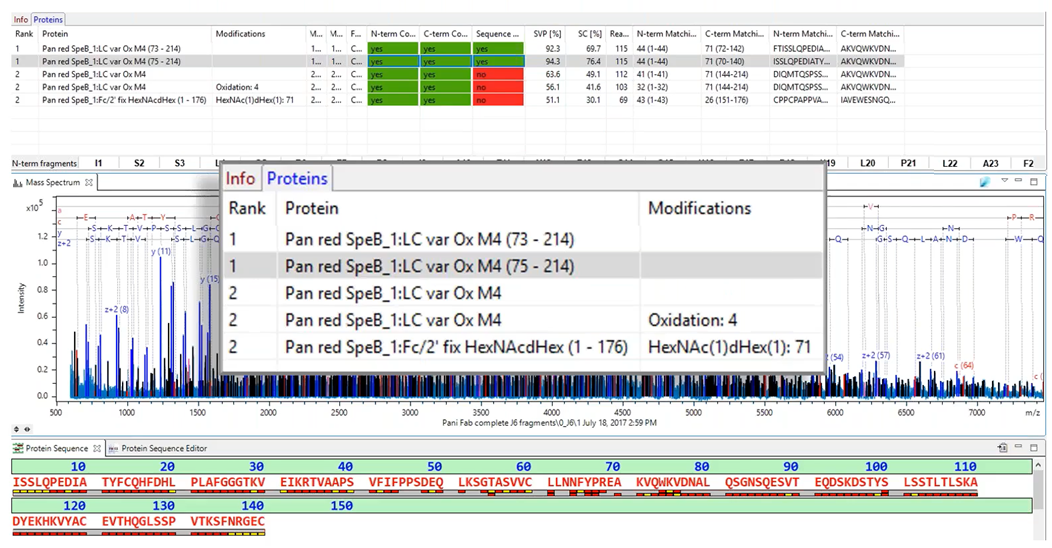
圖11 Panitumumab的抗體蛋白質裁截分析,結果自動導出相關副產物
結語
Bruker除了戮力發展質譜硬體,在軟體、生物資訊大數據、資料庫比對與數據可視化等各方面持續推陳出新,務求提供使用者工作流程自動化的整合性解決方案,並能符合基礎研究或是嚴格的法規需求。SCiLS、MetaboScape與BioPharma Compass的更新,足見跨越病理、表型與代謝、生物製藥等各領域的應用,各軟體之間更能自由導入或導出數據,為使用者提供資料處理的彈性與自由度。
參考文獻:
- Joumbou-Feunang et al. BioTransformer: a comprehensive computational tool for small molecule metabolism prediction and metabolite identification, Journal of Cheminformatics, 2019, 5;11(1):2.
- Fredette, J. and Diehl, D. Meeting Analytical Demands for Biopharmaceuticals with Mass Spectrometry in Late Development, Manufacturing, and Quality Control. The Column. 2016, 12, 10-13.
- http://www.pharmtech.com/therapeutic-product-rd-market-trends, sept 2017
- Jones J, Pack L, Hunter JH, Valliere-Douglass JF. Native size-exclusion chromatography-mass spectrometry: suitability for antibody–drug conjugate drug-to-antibody ratio quantitation across a range of chemotypes and drug-loading levels, mAbs, 2020, 12:1, 1682895.
